2023. 10. 9. 18:10ㆍ개발/💠 Alchemist
1. 분류 모델 중간 정리

2. 부스팅
부스팅
여러개의 약한 분류기를 순차적으로 학습 / 예측하면서 잘못 예측한 데이터에 가중치를 부여해 오류를 개선해 나가면서 학습하는 방식
2-1. GBM ( Gradient Boosting Machine )
GBM의 정의 및 특징
가장 기본적인 부스팅으로, 경사 하강법을 이용해 가중치 업데이트를 수행한다

h(x) : 오류값, y : 실제값, x : 데이터의 피처, F(x) : 예측값 이라고 할 때
h(x) = y - F(x)
h(x)가 최솟값을 갖도록 가중치 설정
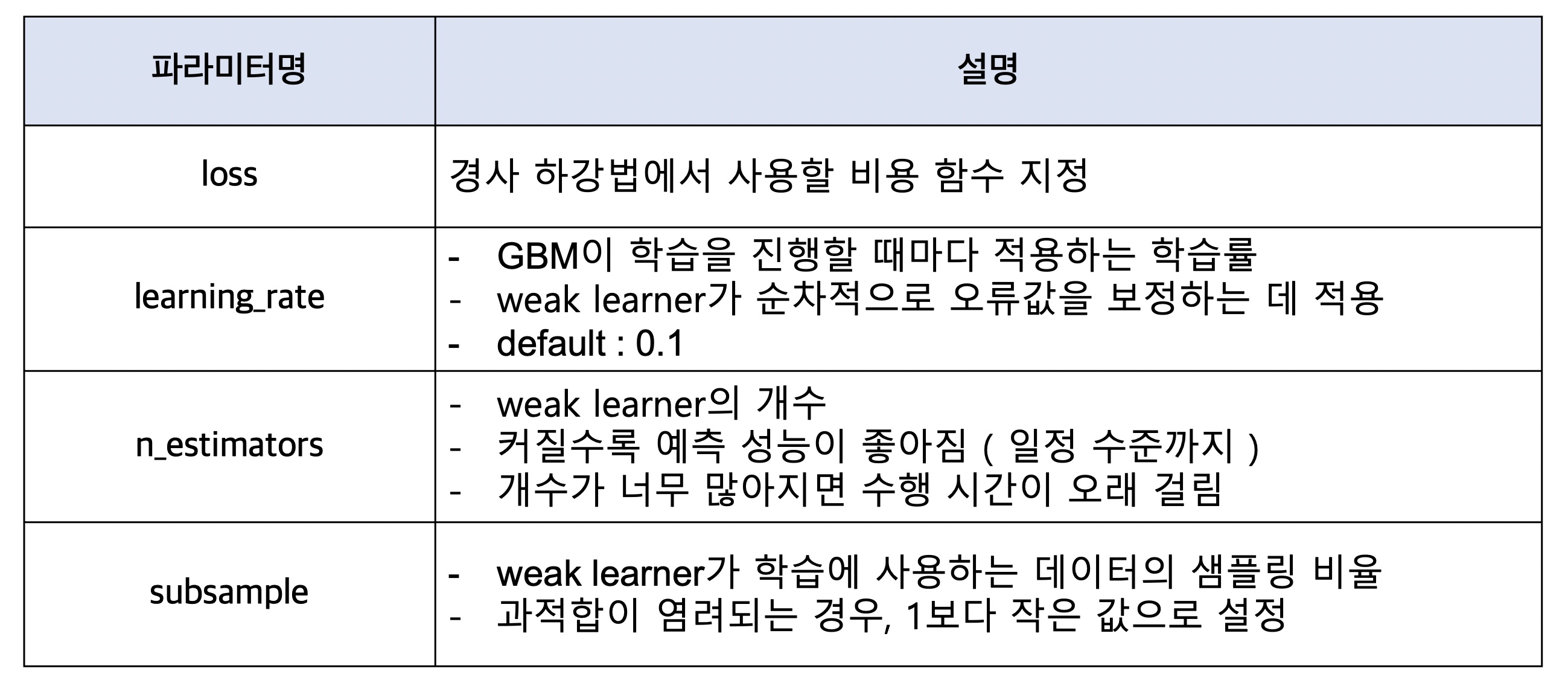
GBM 파라미터
2-2. XGBoost ( eXtra Gradient Boost )
XGBoost의 정의 및 특징
GBM에 기반해서 단점을 보완한 부스팅이며 분류에 있어서 뛰어난 예측 성능을 나타내는 알고리즘이다
XGBoost의 주요 장점
- 분류와 회귀 영역에서 뛰어난 예측 성능
- 병렬 수행 등 다양한 기능으로 GBM에 비해 빠른 수행 속도
- 과적합 규제 기능
- 나무 가지치기 : 긍정 이득이 없는 분할 줄임
- 조기 중단 : 최적화된 반복 수행 횟수를 채웠거나 최적화가 이뤄지면 반복 중단
- 결측치 자체 처리
XGBoost의 구현 패키지 - 파이썬 래퍼, 사이킷런 래퍼
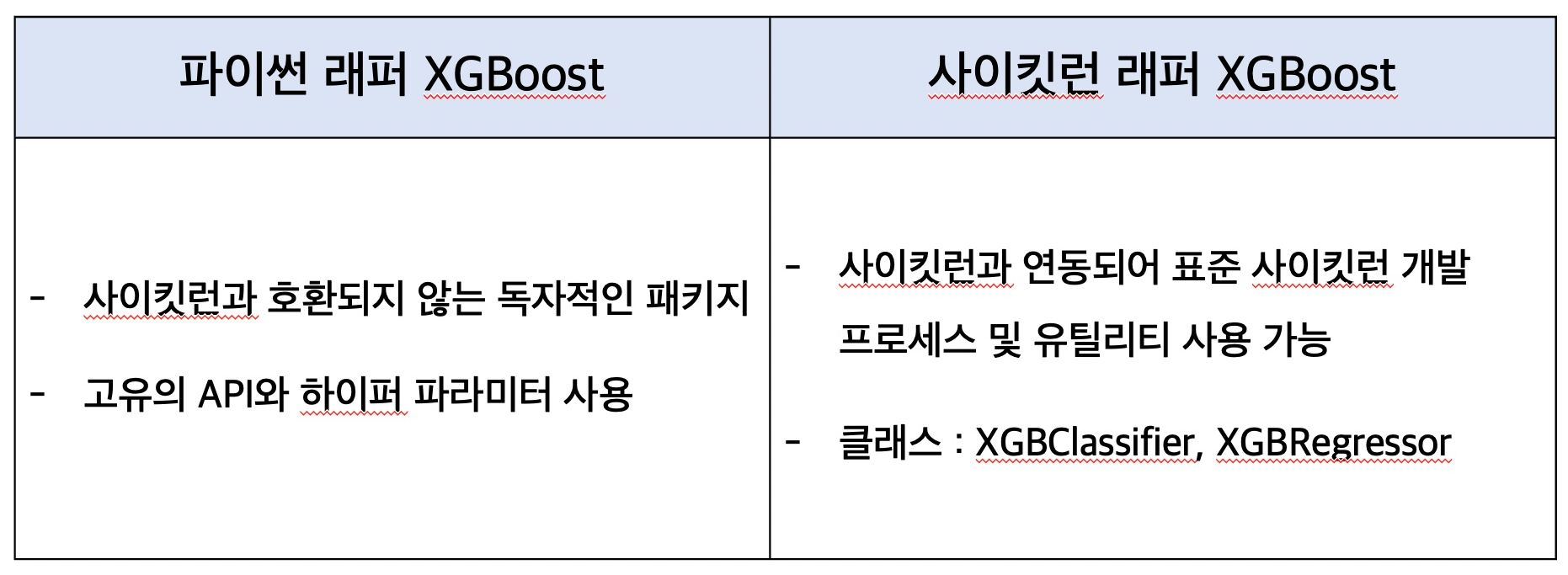
파이썬 래퍼 XGBoost 파라미터 정리
- 일반 파라미터 : 스레드의 개수나 silent 모드 등 선택, 일반적으로 default값 유지
- 부스터 파라미터 : 트리 최적화, 부스팅, regularization 조정
- 학습 태스크 파라미터 : 학습에 사용되는 객체 함수, 평가 지표
1 ) 일반 파라미터

2 ) 부스터 파라미터


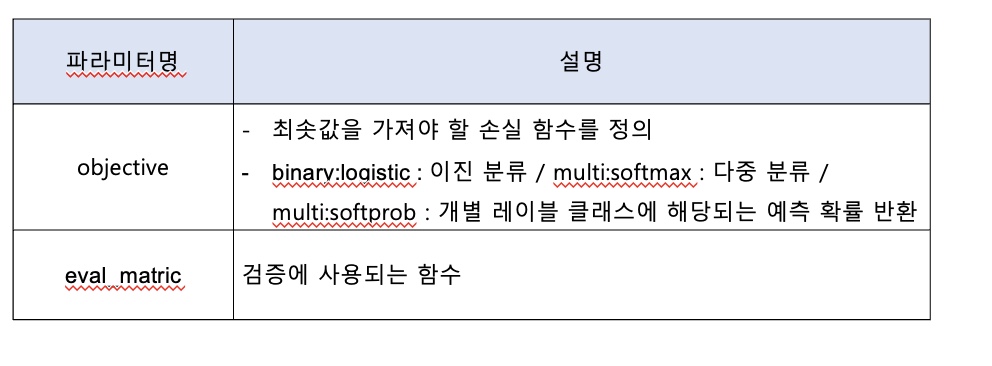
3 ) 학습 태스크 파라미터
4 ) 최적 파라미터 도출 - cv() api

적용 - 위스콘신 유방암 예측
데이터 세트 로딩 및 레이블 칼럼 초기화, 정보 확인
import xgboost as xgb
from xgboost import plot_importance
import pandas as pd
import numpy as np
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
import warnings
warnings.filterwarnings('ignore')
dataset = load_breast_cancer()
features = dataset.data
labels = dataset.target
cancer_df = pd.DataFrame(data=features, columns=dataset.feature_names)
cancer_df['target'] = labels
cancer_df.head(3)
print(dataset.target_names)
print(cancer_df['target'].value_counts())
학습 / 테스트 데이터 분리
X_features = cancer_df.iloc[:, :-1]
y_label = cancer_df.iloc[:, -1]
# 전체 데이터 중 80%는 학습용 데이터, 20%는 테스트용 데이터 추출
X_train, X_test, y_train, y_test = train_test_split(X_features, y_label, test_size = 0.2, random_state = 156)
# 위에서 만든 X_train, y_train을 다시 쪼개서 90%는 학습과 10%는 검증용 데이터로 분리 -> XGBoost에서 제공하는 검증 성능 평가와 조기 중단을 수행해보려고
X_tr, X_val, y_tr, y_val = train_test_split(X_train, y_train, test_size=0.1, random_state=156)
print(X_train.shape, X_test.shape)
print(X_tr.shape, X_val.shape)
학습용, 검증용, 테스트용 데이터를 DMatrix로 생성하기
- 파이썬 래퍼 XGBoost는 XGBoost만의 전용 데이터 객체인 DMatrix를 사용한다
- DMatrix의 파라미터 : data(피처 데이터 세트) / label(레이블 데이터 세트)
dtr = xgb.DMatrix(data=X_tr, label=y_tr)
dval = xgb.DMatrix(data=X_val, label=y_val)
dtest = xgb.DMatrix(data=X_test, label=y_test)
XGBoost 모델 학습 / 예측
# 파이썬 래퍼 XGBoost의 학습 함수, train()의 파라미터 정리
## eval_metric : 조기 중단을 위한 성능 지표 (낮을수록 성능이 좋다)
params = {
'max_depth':3,
'eta':0.05,
'objective':'binary:logistic',
'eval_metric':'logloss'
}
num_rounds = 400
# 평가용 데이터 세트 - 튜플 형식으로 학습용 데이터 세트와 평가용 데이터 세트를 'train', 'eval'로 명시하여 표기
eval_list = [(dtr,'train'),(dval,'eval')] #[(dval,'eval')]만 표기해도 됨
# XGBoost 모델 학습
## num_boost_round : 부스팅 몇 번 수행할지
## early_stopping_rounds : 조기 중단할 수 있는 최소 반복 횟수
## evals : 조기 중단을 위한 평가용 데이터 세트
xgb_model = xgb.train(params=params, dtrain=dtr, num_boost_round=num_rounds, \
early_stopping_rounds=50, evals=eval_list)
# XGBoost 모델 예측
pred_probs = xgb_model.predict(dtest)
print('predict() 수행 결괏값을 10개만 표시, 예측 확률 값으로 표시됨')
print(np.round(pred_probs[:10],3))
# 파이썬 래퍼 XGBoost의 predict()은 예측 확률을 표시하므로 0.5이하면 0, 초과면 1로 결정하는 로직 추가
preds = [1 if x > 0.5 else 0 for x in pred_probs]
print('예측값 10개만 표시:',preds[:10])
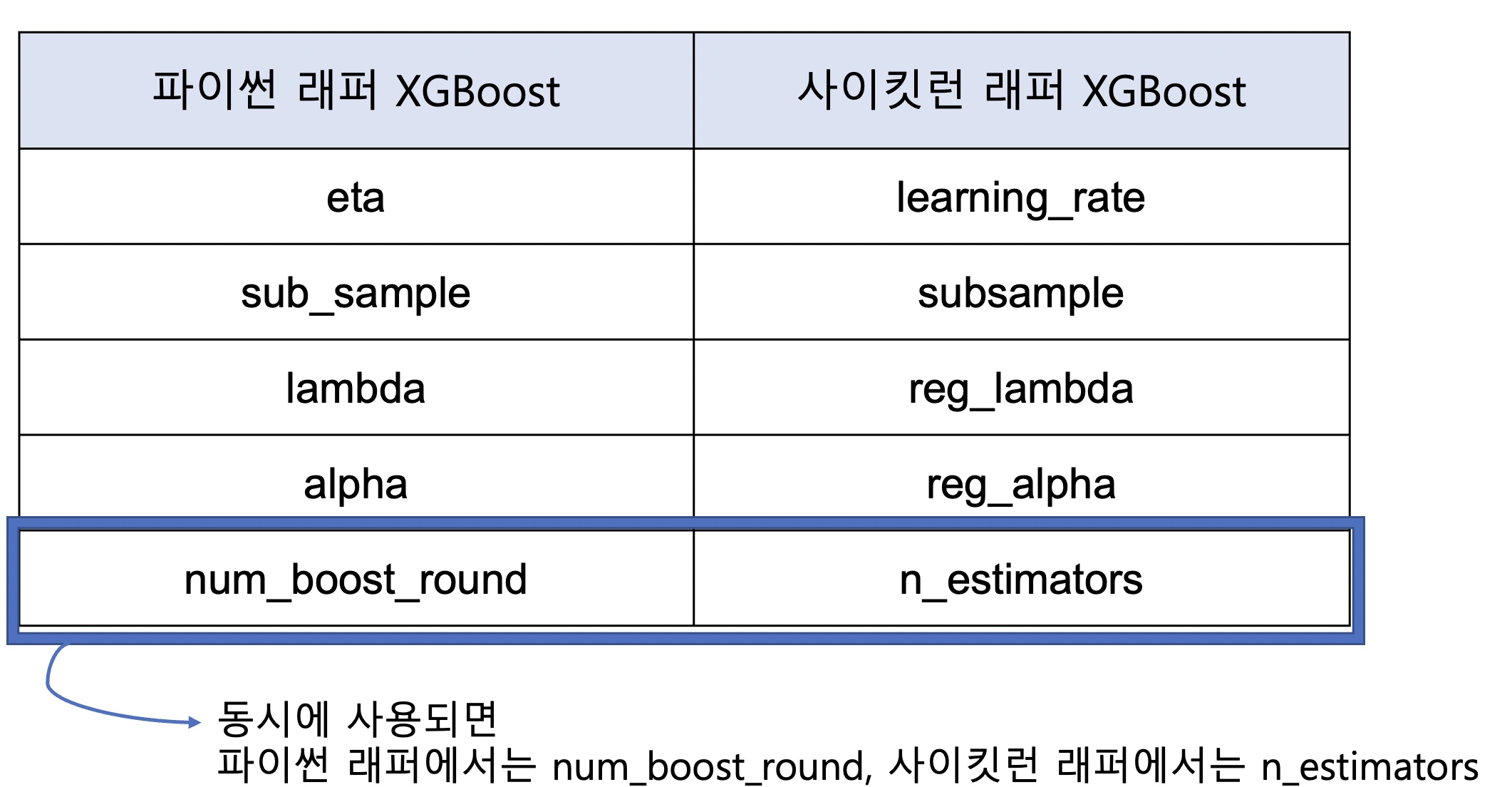
사이킷런 래퍼 XGBoost 파라미터 정리 (파이썬 래퍼와 비교)
조기 중단 파라미터

적용 - 위스콘신 유방암 예측 ( 파이썬 래퍼 XGBoost 코드에 이어서 )
# 사이킷런 래퍼 XGBoost import
from xgboost import XGBClassifier
# XGBoost 객체 생성
xgb_wrapper = XGBClassifier(n_estimators=400, learning_rate=0.05, max_depth=3)
# 조기 중단을 위한 성능 평가 데이터 세트
evals = [(X_tr, y_tr),(X_val, y_val)]
# 모델 학습 - 조기 중단을 위한 최소 반복 횟수 50으로 설정
xgb_wrapper.fit(X_tr, y_tr, early_stopping_rounds=50, eval_metric="logloss", eval_set=evals, verbose=True)
# 모델 예측
ws50_preds = xgb_wrapper.predict(X_test)
ws50_pred_proba = xgb_wrapper.predict_proba(X_test)[:, 1]
조기 중단 전 최소 반복 횟수 10으로 설정했을 때
xgb_wrapper.fit(X_tr, y_tr, early_stopping_rounds=10, eval_metric='logloss',eval_set=evals, verbose=True)
ws10_preds = xgb_wrapper.predict(X_test)
ws10_pred_proba = xgb_wrapper.predict_proba(X_test)[:, 1]조기 중단 전 최소 반복 횟수를 50으로 설정했을 때와 10으로 설정했을 때의 성능을 평가한 결과
50으로 설정했을 때의 성능이 더 좋게 나왔다
조기 중단 전 최소 반복 횟수를 너무 낮추게 되면 아직 성능이 향상될 여지가 남아있음에도 불구하고
10번의 반복 내에 성능이 향상되지 않으면 바로 중단을 시켜버리기 때문에
오히려 충분한 학습이 이루어지지 않아 예측 성능이 낮아질 수 있다
2-3. LightGBM
LightGBM의 정의 및 특징
최근에 가장 각광받고 있는 부스팅 알고리즘, XGBoost의 느린 수행 시간이 보완됨
LightGBM의 주요 장점 (XGBoost와 비교)
- 빠른 학습과 예측 수행, 더 작은 메모리 사용
- XGBoost와 예측 성능은 비슷하지만 기능상 더 다양함
- 카테고리형 피처의 자동 변환과 최적 분할
- 리프 중심 트리 분할 ( Leaf Wise ) : 트리의 균형을 맞추는 대신 최대 손실 값을 가지는 리프 노드를 지속적으로 분할하면서 트리가 깊어지고 비대칭적인 규칙 트리 생성
* 리프 중심 트리 분할 방식을 사용하여 수행 시간을 훨씬 줄일 수 있지만 과적합의 가능성을 높인다는 단점이 있다
LightGBM의 주요 파라미터
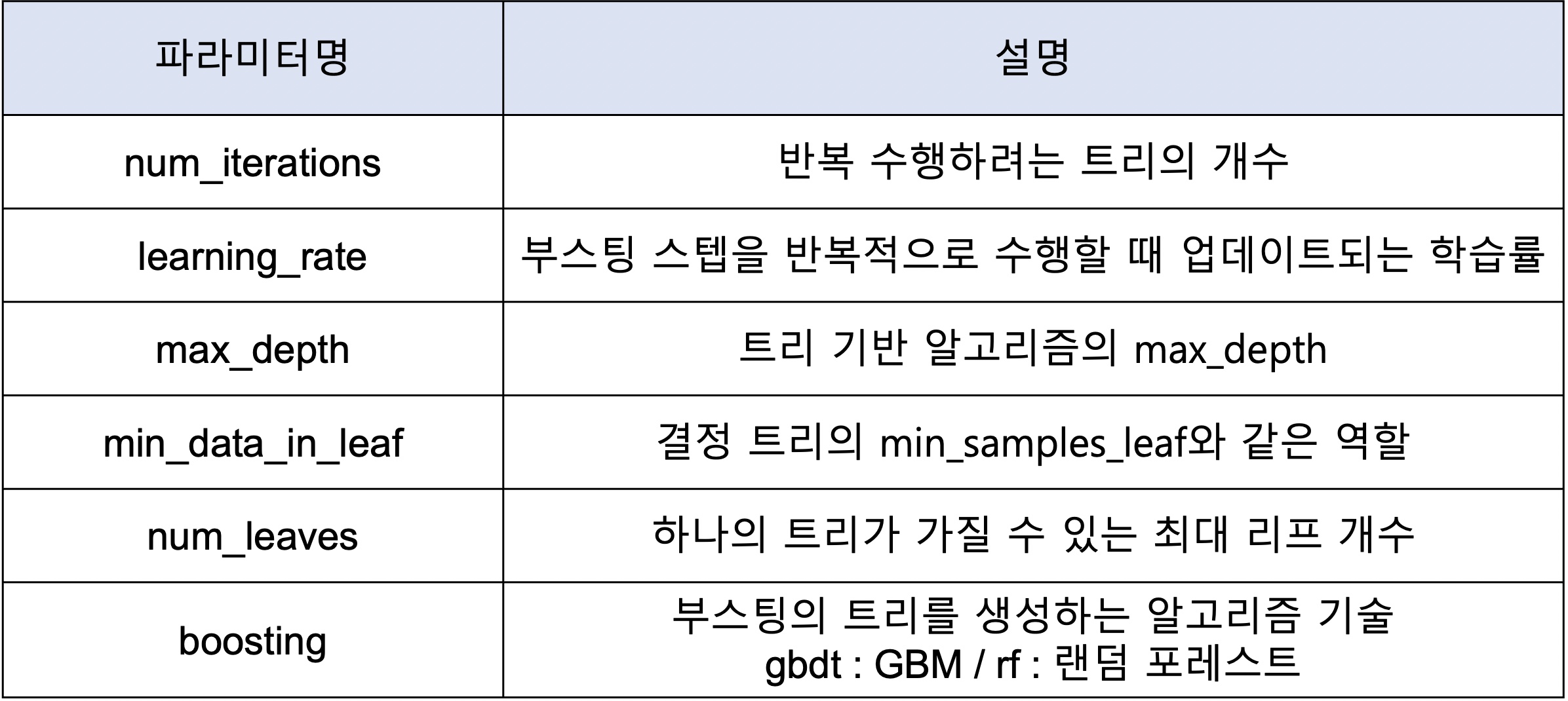

학습 태스크 파라미터
objective : 최솟값을 가져야 할 손실 함수 정의
LightGBM의 파라미터 튜닝 방안
LGBM은 비대칭 트리를 기반으로 하기 때문에 과적합이 될 위험성이 높으므로 튜닝을 통해 이를 제어해줘야 한다
- num_leaves의 개수를 높이면 정확도가 높아지지만 반대로 트리의 깊이가 깊어지고 모델의 복잡도가 커져 과적합이 될 가능성이 높아진다
- min_data_in_leaf는 num_leaves와 학습 데이터의 크기에 따라 달라지지만 보통 큰 값으로 설정하면 트리가 깊어지는 것을 방지한다
- max_depth는 트리의 깊이를 제한한다
적용 - 위스콘신 유방암 예측
from lightgbm import LGBMClassifier
import pandas as pd
import numpy as np
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
dataset = load_breast_cancer()
cancer_df = pd.DataFrame(data=dataset.data, columns=dataset.feature_names)
cancer_df['target'] = dataset.target
X_features = cancer_df.iloc[:,:-1]
y_label = cancer_df.iloc[:, -1]
X_train, X_test, y_train, y_test = train_test_split(X_features, y_label, test_size=0.2, random_state=156)
X_tr, X_val, y_tr, y_val = train_test_split(X_train, y_train, test_size=0.1, random_state=156)
# LGBM 객체 생성
lgbm_wrapper = LGBMClassifier(n_estimators=400, learning_rate=0.05)
# 성능 평가용 데이터 세트
evals=[(X_tr, y_tr),(X_val, y_val)]
# 모델 학습
lgbm_wrapper.fit(X_tr, y_tr, early_stopping_rounds=50, eval_metric="logloss", eval_set=evals, verbose=True)
# 모델 예측
preds=lgbm_wrapper.predict(X_test)
pred_proba=lgbm_wrapper.predict_proba(X_test)[:, 1]
3. 베이지안 최적화 기반의 HyperOpt를 이용한 하이퍼 파라미터 튜닝
부스팅 알고리즘 모델들(GBM, XGBoost, LGBM)은 기본적으로 모두 파라미터의 개수가 매우 많다
따라서 하이퍼 파라미터 튜닝이 필요하고
주로 사용되는 방법은 베이지안 최적화를 기반으로 한 HyperOpt를 적용하는 방식이다
베이지안 최적화
베이지안 최적화는 목적 함수식을 제대로 알지 못하는 블랙 박스 형태의 함수에서
최대 또는 최소 반환값을 만드는 최적 입력값을 찾아야 할 때 유용하게 쓰인다
베이지안 최적화는 획득 함수와 대체 모델로 구성되어 있으며 아래와 같이 작동한다
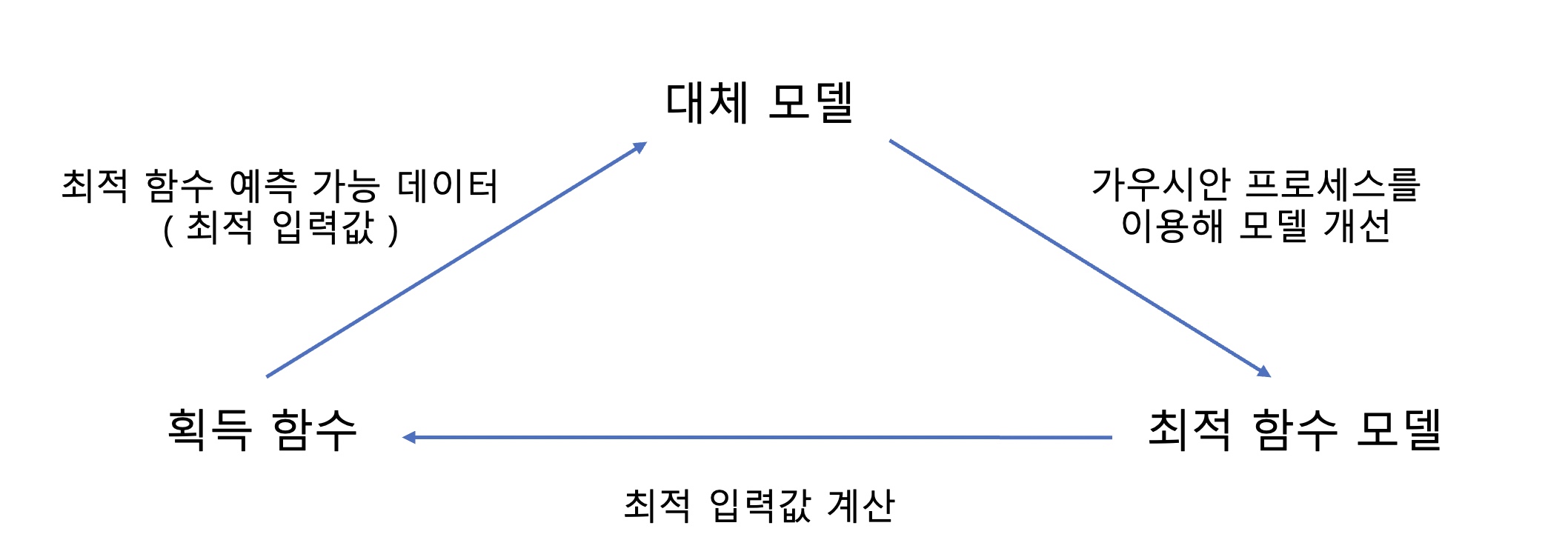
획득함수가 대체 모델에게 최적 입력값(최적 함수를 예측하기 위한 데이터)을 제공하면
대체 모델은 최적 입력값을 바탕으로 가우시안 프로세스를 이용해 모델을 개선시킨다
즉, 획득함수가 제공한 최적 입력값을 이용해 최적 함수 모델을 업데이트한다
업데이트된 최적 함수 모델을 기반으로 획득함수는 더 정확한 입력값을 계산할 수 있게 된다
그림과 함께 구체적으로 설명해보자면 아래와 같다

초기에는 랜덤하게 하이퍼 파라미터들을 샘플링하고 성능 결과를 관측한다
위의 그림에서 주황색 점선이 베이지안 최적화를 통해 구해내야 하는 최적 함수 모델이며
검정색 점은 랜덤하게 입력된 하이퍼 파라미터를 기반으로 관측된 성능 지표 결괏값을 의미한다
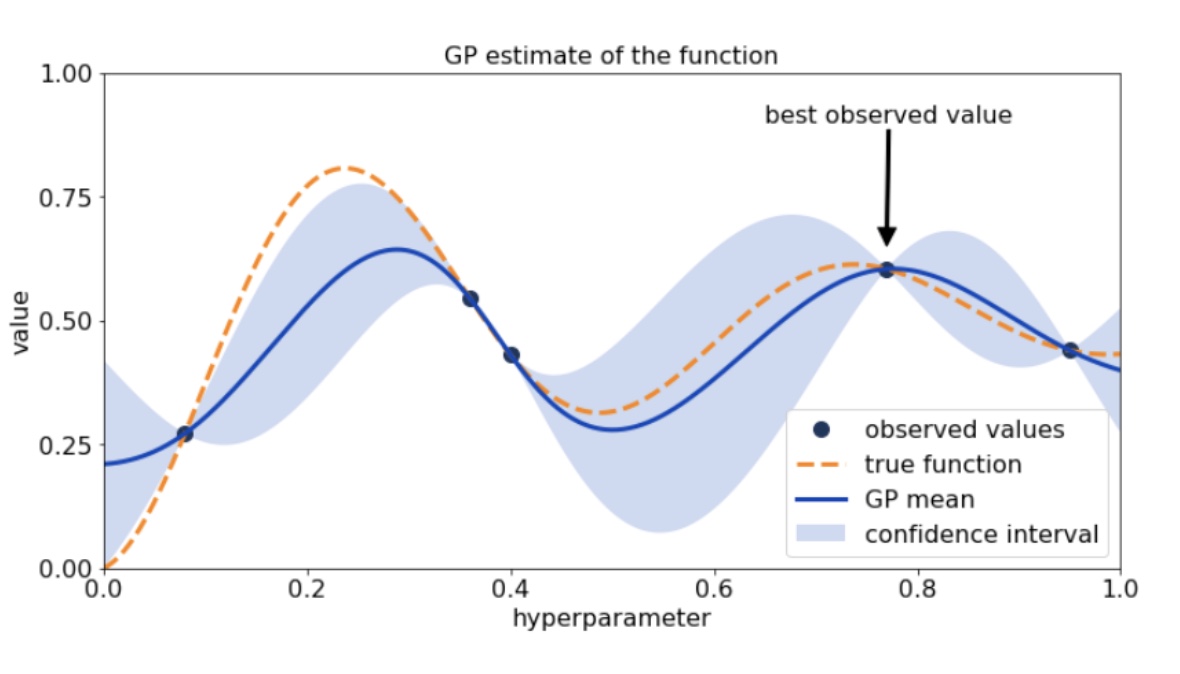
관측된 값을 기반으로 대체 모델은 최적 함수를 추정한다
위의 그림에서 파란색 실선이 대체 모델이 추정한 최적 함수이며
연한 파란색으로 표현된 구간은 결괏값의 오류 편차, 즉 추정 함수의 불확실성을 의미한다
최적 관측값은 y축 value에서 가장 높은 값을 가질 때의 하이퍼 파라미터다

획득 함수는 다음으로 관측할 하이퍼 파라미터 값을 계산한다
획득 함수는 이전 최적 관측값보다 더 큰 최댓값을 가질 가능성이 높은 지점을 찾아서 대체 모델에 전달한다
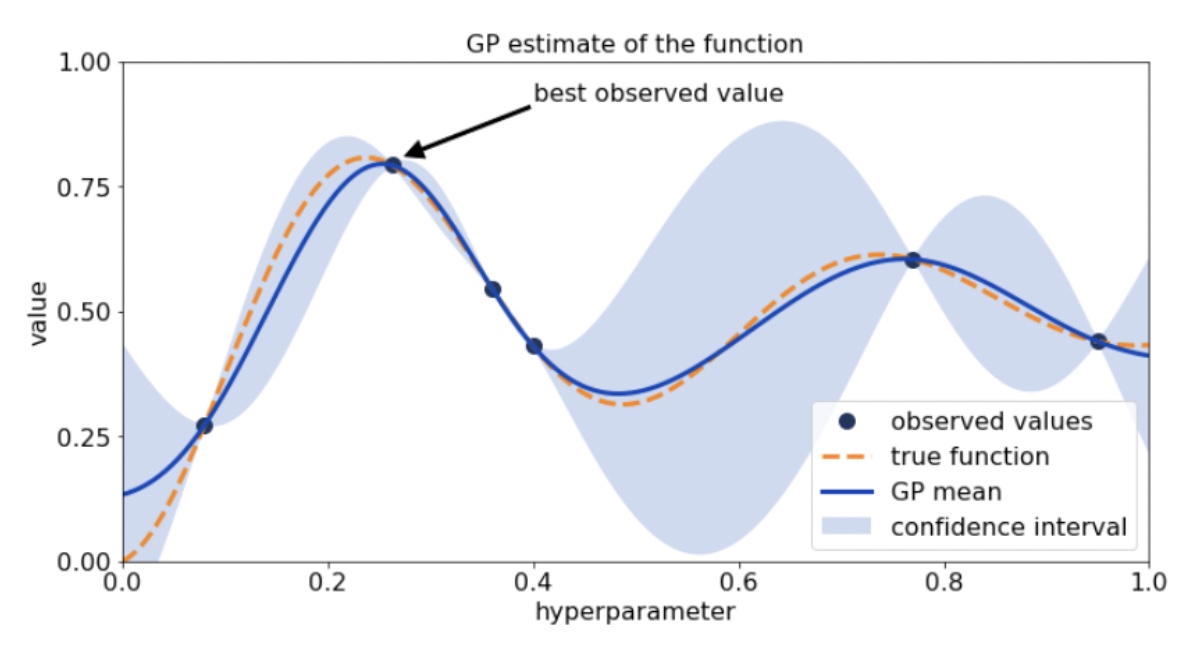
획득 함수로부터 전달된 하이퍼 파라미터를 수행하여
관측된 값을 기반으로 대체 모델은 갱신되어 다시 최적 함수를 예측한다
2번째 그림과 비교해보았을 때 성능 지표 결괏값이 최대인 지점( 0.25 부근 )에서의 오류 편차값이 줄어든 것을 확인할 수 있다
HyperOpt
HyperOpt : 베이지안 최적화를 하이퍼 파라미터 튜닝에 적용할 수 있도록 제공되는 파이썬 패키지
- 대체 모델이 최적 함수를 추정할 때 가우시안 프로세스가 아닌 트리 파르젠을 사용
- 목적 함수 반환값의 최솟값을 가지는 최적 입력값 유추
HyperOpt 활용 로직
입력 변수명과 입력값의 검색 공간 설정
-> 목적 함수 설정
-> 목적 함수의 반환값이 최솟값을 가지도록 하는 최적 입력값 유추
1 ) 입력 변수명과 입력값의 검색 공간 설정 - 파라미터 이용
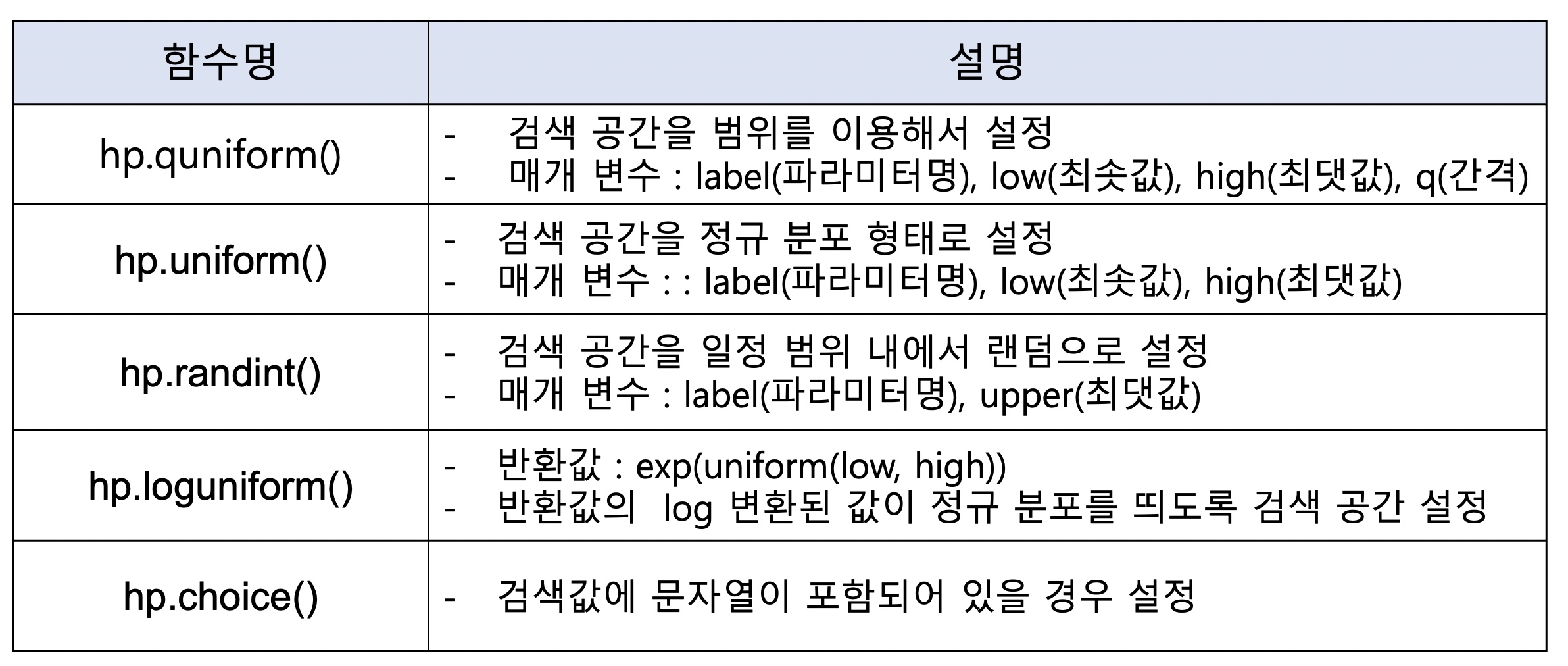
2 ) 목적 함수 설정
- 인자(매개변수) : 하이퍼 파라미터와 검색 공간을 가지는 딕셔너리
- 반환값 : 부스팅 알고리즘을 학습한 후에 관측된 예측 성능 결과

3 ) 목적 함수의 반환값이 최솟값을 가지도록 하는 최적 입력값 유추
- fmin() 함수 이용
fmin() 함수 파라미터 정리

주의 사항
- HyperOpt의 입력값과 반환값은 모두 실수형이기 때문에 형변환을 잘 해줘야 한다
- 성능 값이 클수록, 좋은 성능 지표일 경우 HyperOpt의 목적 함수의 반환값에 -1을 곱해줘야 한다
적용 - 위스콘신 유방암 예측
기본 세팅 - import, 데이터세트 로드, 학습/테스트 데이터 분리, 평가용 데이터 세트 설정
import xgboost as xgb
from xgboost import plot_importance
import pandas as pd
import numpy as np
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
import warnings
warnings.filterwarnings('ignore')
dataset = load_breast_cancer()
features = dataset.data
labels = dataset.target
cancer_df = pd.DataFrame(data=features, columns=dataset.feature_names)
cancer_df['target'] = labels
X_features = cancer_df.iloc[:, :-1]
y_label = cancer_df.iloc[:, -1]
X_train, X_test, y_train, y_test = train_test_split(X_features, y_label, test_size = 0.2, random_state = 156)
X_tr, X_val, y_tr, y_val = train_test_split(X_train, y_train, test_size=0.1, random_state=156)
검색 공간 설정
from hyperopt import hp
xgb_search_space = {
'max_depth':hp.quniform('max_depth',5,20,1),
'min_child_weight':hp.quniform('min_child_weight',1,2,1),
'learning_rate':hp.uniform('learning_rate',0.01,0.2),
'colsample_bytree':hp.uniform('colsample_bytree',0.5,1),
}목적 함수 설정
from sklearn.model_selection import cross_val_score
from xgboost import XGBClassifier
from hyperopt import STATUS_OK
# 목적함수 설정
## 인자 : 검색 공간
def objective_func(search_space):
xgb_clf = XGBClassifier(n_estimators=100, max_depth=int(search_space['max_depth']), min_child_weight=int(search_space['min_child_weight']),
learning_rate=search_space['learning_rate'], colsample_bytree=search_space['colsample_bytree'], eval_metric='logloss')
accuracy=cross_val_score(xgb_clf, X_train, y_train, scoring='accuracy',cv=3)
# 목적 함수 반환값 : 성능 평가 지표(accuracy)
# fmin()은 최솟값을 최적값으로 반환하기 때문에 -1 곱해서 반환
return {'loss':-1*np.mean(accuracy),'status':STATUS_OK}
hyperopt 이용한 최적 하이퍼 파라미터 계산
from hyperopt import fmin, tpe, Trials
trial_val = Trials()
best = fmin(fn=objective_func,
space=xgb_search_space,
algo=tpe.suggest,
max_evals=50,
trials=trial_val, rstate=np.random.default_rng(seed=9))
print('best:',best)
4. 스택킹 ( Stacking )
스택킹의 정의 및 특징
- 개별 모델로 예측한 결과를 기반으로 다시 최종 예측을 수행하는 방식
- 메타 모델 : 여러 개별 모델들의 예측 결과를 다시 기반으로 하여 학습 / 예측하는 방식

스택킹 앙상블 과정
- 여러 개별 모델이 학습한 뒤 예측 결과 도출
- 개별 모델의 예측 결과들을 stacking 형태로 결합해서 최종 메타 모델의 입력으로 사용

적용 - 위스콘신 유방암 예측
기본 세팅
import numpy as np
from sklearn.neighbors import KNeighborsClassifier
from sklearn.ensemble import RandomForestClassifier
from sklearn.ensemble import AdaBoostClassifier
from sklearn.tree import DecisionTreeClassifier
from sklearn.linear_model import LogisticRegression
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score
cancer_data=load_breast_cancer()
X_data = cancer_data.data
y_label = cancer_data.target
X_train, X_test, y_train, y_test = train_test_split(X_data, y_label, test_size=0.2, random_state=0)
개별 분류기 생성 및 학습 / 예측
# 개별 분류기 생성
knn_clf = KNeighborsClassifier(n_neighbors=4)
rf_clf = RandomForestClassifier(n_estimators=100, random_state=0)
dt_clf = DecisionTreeClassifier()
ada_clf = AdaBoostClassifier(n_estimators=100)
# 최종 메타 모델(분류기) 생성
lr_final = LogisticRegression()
# 개별 분류기 학습
knn_clf.fit(X_train, y_train)
rf_clf.fit(X_train, y_train)
dt_clf.fit(X_train, y_train)
ada_clf.fit(X_train, y_train)
# 개별 분류기 예측
knn_pred = knn_clf.predict(X_test)
rf_pred = rf_clf.predict(X_test)
dt_pred = dt_clf.predict(X_test)
ada_pred = ada_clf.predict(X_test)
개별 분류기의 예측 결과 stacking -> 최종 학습 데이터
pred = np.array([knn_pred, rf_pred, dt_pred, ada_pred])
print(pred.shape)
pred = np.transpose(pred)
print(pred.shape)
위에서 생성한 최종 학습 데이터 최종 메타 모델에 학습시킨 뒤 예측 수행 -> 평가
lr_final.fit(pred, y_test)
final = lr_final.predict(pred)
print('최종 메타 모델의 예측 정확도: {0:.4f}'.format(accuracy_score(y_test, final)))
CV set 기반 Stacking
step 1 : 개별 base 모델 학습, 예측값 도출

- 학습 데이터를 K개의 fold로 나눔
- ( K-1 )개의 fold를 학습 데이터로 하여 base 모델 학습 (K번 반복)
* 검증 fold 1개를 예측한 결과 (K fold) -> 최종 메타 모델의 학습 데이터
* 테스트 데이터를 예측한 결과의 평균 -> 최종 메타 모델의 테스트 데이터
step 2 : 최종 메타 모델 학습

- 각 base 모델이 생성한 학습용 데이터를 stacking -> 최종 메타 모델의 학습 데이터 세트
- 각 base 모델이 생성한 테스트용 데이터를 stacking -> 최종 메타 모델의 테스트 데이터 세트
- 최종 학습용 데이터 + 원본 학습 레이블 데이터로 학습
- 최종 테스트용 데이터로 예측 -> 원본 테스트 레이블 데이터로 평가
적용 - 위스콘신 유방암 예측
CV set 기반 Stacking - step1
from sklearn.model_selection import KFold
from sklearn.metrics import mean_absolute_error
def get_stacking_base_datasets(model, X_train_n, y_train_n, X_test_n, n_folds):
# 학습 데이터 -> n개 폴드로 분리
kf = KFold(n_splits=n_folds, shuffle=False)
train_fold_pred=np.zeros((X_train_n.shape[0],1))
test_pred=np.zeros((X_test_n.shape[0], n_folds))
print(model.__class__.__name__, 'model 시작')
# n번 반복
for folder_counter, (train_index, valid_index) in enumerate(kf.split(X_train_n)):
print('\t폴드 세트: ',folder_counter,' 시작 ')
# n개로 분리된 데이터를 학습 데이터와 테스트 데이터로 분리
X_tr = X_train_n[train_index]
y_tr = y_train_n[train_index]
X_te = X_train_n[valid_index]
# 학습 데이터로 모델 학습
model.fit(X_tr, y_tr)
# 테스트 데이터로 모델 예측
train_fold_pred[valid_index, :] = model.predict(X_te).reshape(-1, 1)
test_pred[:, folder_counter]=model.predict(X_test_n)
# 예측 데이터의 평균 내기
test_pred_mean = np.mean(test_pred, axis=1).reshape(-1,1)
return train_fold_pred, test_pred_mean
# 개별 분류기 step1 실시
knn_train, knn_test = get_stacking_base_datasets(knn_clf, X_train, y_train, X_test, 7)
rf_train, rf_test = get_stacking_base_datasets(rf_clf, X_train, y_train, X_test, 7)
dt_train, dt_test = get_stacking_base_datasets(dt_clf, X_train, y_train, X_test, 7)
ada_train, ada_test = get_stacking_base_datasets(ada_clf, X_train, y_train, X_test, 7)
CV set 기반 Stacking - step2
# 학습 데이터 stacking
Stack_final_X_train = np.concatenate((knn_train, rf_train, dt_train, ada_train), axis = 1)
# 테스트 데이터 stacking
Stack_final_X_test = np.concatenate((knn_test, rf_test, dt_test, ada_test), axis = 1)
print('원본 학습 피처 데이터 shape:', X_train.shape, '원본 테스트 피처 shape:',X_test.shape)
print('스태킹 학습 피처 데이터 shape:',Stack_final_X_train.shape,
'스태킹 테스트 피처 데이터 shape:',Stack_final_X_test.shape)
최종 메타 모델 학습 및 예측 -> 평가
lr_final.fit(Stack_final_X_train, y_train)
stack_final = lr_final.predict(Stack_final_X_test)
print('최종 메타 모델의 예측 정확도: {0:.4f}'.format(accuracy_score(y_test, stack_final)))'개발 > 💠 Alchemist' 카테고리의 다른 글
| 💠 분류 - 캐글 산탄데르 고객 만족 예측 💠 (0) | 2023.10.31 |
|---|---|
| 💠 AIchemist 4주차 💠 (0) | 2023.10.31 |
| 💠 Alchemist 3주차 💠 (0) | 2023.10.04 |
| 💠 머신러닝 분류 모델 ( 1 ) 💠 (0) | 2023.10.03 |
| 💠 Alchemist 2주차 💠 (0) | 2023.10.01 |